Alinhamentos Múltiplos e Árvores Filogenéticas¶
O alinhamento múltiplo consiste no alinhamento de 3 ou mais sequências de DNA, RNA ou proteínas. Por norma, as sequências relacionam-se evolutivamente, partilhando uma linhagem e um ancestral comum. A partir do alinhamento torna-se possível inferir a homlogia entre as várias sequências e proceder com uma análise filogenética, por forma a identificar situações de evolução distintas ou semelhantes entre genes.
Neste trabalho, escolhemos 4 proteínas hipotéticas, que fazem parte da zona do genoma em estudo, para analisar por alinhamento múltiplo e árvores filogenéticas na ferramenta Clustal Omega (http://www.ebi.ac.uk/Tools/msa/clustalo/). O alinhamento foi feito apenas com o top 10 dos resultados do blast, que correspondem às proteínas com maior homologia relativamente à proteína em estudo. Estas proteínas foram escolhidas tendo em conta os seguintes critérios:
Não possuem função conhecida;
Os resultados do blast consistem em algumas proteinas com função conhecida e outras não;
Identificamos o nome, accession number e descricao dos domínios que possuem.
O segundo critério foi considerado, uma vez que se a nossa proteína tiver um ancestral comum com aquelas que possuem função, é possível que também a desempenhe e a inclusão daquelas que não têm função associada permite perceber a relação evolutiva entre as diversas proteínas.
lpg0268¶
Para esta proteína, apenas 3 proteínas que fazem parte dos resultados do blast (top 10) possuem função identificada (A0A140J104, A0A129N4B5, A0A128KV86), nomeadamente atividade hidrolítica, e pertencem a susbspécies ou estirpes diferentes da Legionella. A proteína lpg0268 possui o domínio HD_3, por norma associado à função de fosfohidrolase metalo-dependente.
Analisando os resultados do alinhamento múltiplo podemos ver, como de esperar pelos resultados do blast, que as sequências são bastante semelhantes entre si. A proteína em estudo não apresenta uma relação direta na árvore filogenética com nenhuma das três proteínas que apresentam função conhecida. Contudo, a proteína A0A140J104 pode ser considerada o ancestral comum da nossa proteína e das proteínas A0A129N4B5 e A0A128KV86. Aquela com função mais próxima à proteína em estudo na árvore filogenética é a A0A129N4B5. Tendo em conta a existência de um ancestral comum, o fato de as proteínas pertencerem a diferentes susbpécies ou estirpes do mesmo organismo (o que indica que a relação é mais próxima, bem como as funções desempenhadas) e a existência de um domínio na nossa proteína associado a atividades hydrolase-like, concluímos que é provável que a proteína lpg0268 desempenhe uma atividade de hidrolase.
 Figura 1 - Resultados do alinhamento múltiplo para a proteína lpg0268.
Figura 1 - Resultados do alinhamento múltiplo para a proteína lpg0268.
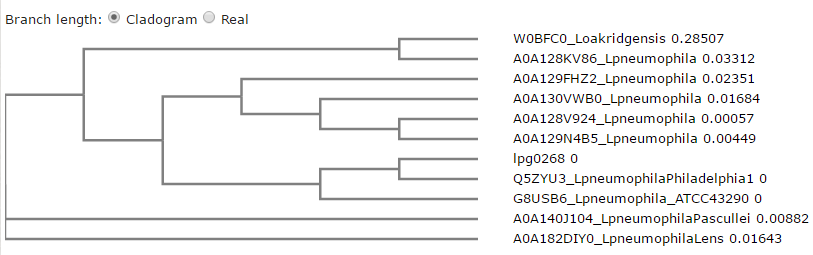 Figura 2 - Árvore filogenética para a proteína lpg0268.
Figura 2 - Árvore filogenética para a proteína lpg0268.
lpg0269¶
Para esta proteína, apenas 4 proteínas que fazem parte dos resultados do blast (top 10) possuem função identificada (A0A128J1H8, A0A182DM20, A0A129FHQ9, A0A128TDJ0), nomeadamente atividade de transaminase, e pertencem a susbspécies ou estirpes diferentes da Legionella. A proteína lpg0269 possui o domínio P-loop_NTPase super family, por norma associado à função de hidrolase contendo um P-loop nucleosídeo trifosfato, e o domínio PI-PLCc_GDPD_SF super family, associado à função de phosphodiesterases (um tipo de hidrolase) PI-PLC-like.
Analisando os resultados do alinhamento múltiplo podemos ver, como de esperar pelos resultados do blast, que as sequências são bastante semelhantes entre si. A proteína em estudo não apresenta, no entanto, uma relação direta na árvore filogenética com nenhuma das quatro proteínas que apresentam função conhecida. De fato, as quatro proteínas encontram-se relativamente distantes da proteína em estudo. As transaminases e as hidrolases, apesar de serem ambas enzimas, desempenham funções diferentes, pertecendo a classes diferentes. Visto que as sequências são homólogas, contudo, podemos supor que as hidrolases e as transaminases diferem apenas nalguns aminoácidos na sua sequência proteica. A presença do domínio já referido na nossa proteína faz com que seja mais provável ela possuir atividade de hidrolase do que de transaminase.


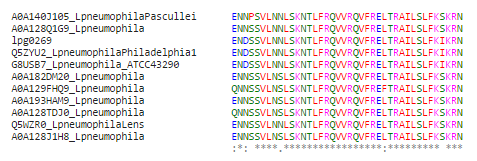 Figura 3 - Resultados do alinhamento múltiplo para a proteína lpg0269.
Figura 3 - Resultados do alinhamento múltiplo para a proteína lpg0269.
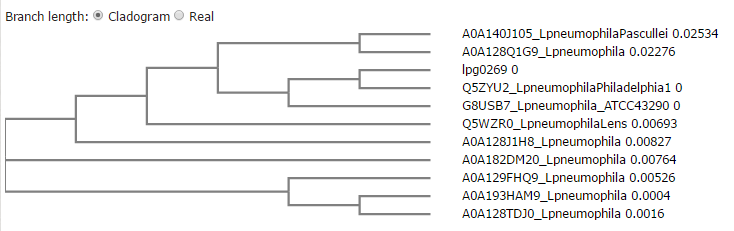 Figura 4 - Árvore filogenética para a proteína lpg0269.
Figura 4 - Árvore filogenética para a proteína lpg0269.
lpg0282¶
Para esta proteína, apenas 7 proteínas que fazem parte dos resultados do blast (top 10) possuem função identificada (A0A182DPP8, A0A128Q2L5, A0A140J121, A0A129FJH3, A0A129F5V7, A0A193HAT7,A0A131NME4), nomeadamente atividade de metiltransferase, e pertencem a susbspécies ou estirpes diferentes da Legionella. A proteína lpg0282 possui o domínio Methyltransf_31, por norma associado à função de metiltransferase.
Analisando os resultados do alinhamento múltiplo podemos ver, como de esperar pelos resultados do blast, que as sequências são bastante semelhantes entre si. A proteína em estudo não apresenta, no entanto, uma relação direta na árvore filogenética com nenhuma das quatro proteínas que apresentam função conhecida. De fato, as proteínas encontram-se relativamente distantes da proteína em estudo. Por análise da árvore é possível observar que a nossa proteína se encontra mais próximo do topo do que as sete proteínascom função, sendo, portanto, o ancestral comum de todas essas. Assim, tendo em conta este fato e também a presença do motivo referido na proteína lpg0282, concluímos que é muito provável que a proteína seja uma metiltransferase.
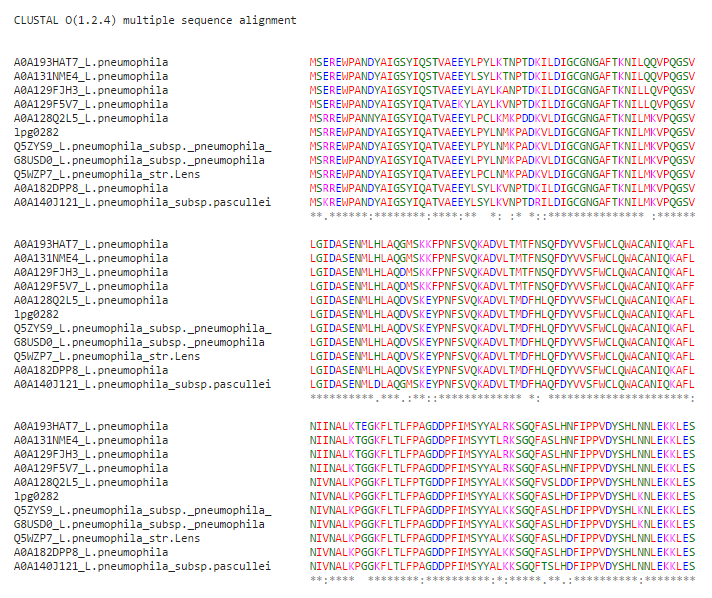
 Figura 5 - Resultados do alinhamento múltiplo para a proteína lpg0282.
Figura 5 - Resultados do alinhamento múltiplo para a proteína lpg0282.
 Figura 6 - Árvore filogenética para a proteína lpg0282.
Figura 6 - Árvore filogenética para a proteína lpg0282.
lpg0428¶
Para esta proteína, apenas 4 proteínas que fazem parte dos resultados do blast (top 10) possuem função identificada (A0A129B501, A0A130Q1T4, A0A129IE10, A0A128SMY7), nomeadamente atividade de liase, e pertencem a susbspécies ou estirpes diferentes da Legionella. A proteína lpg0428 e possui o domínio Glo_EDI_BRP_like, encontrado em metalloproteinas, as quais são, por norma, enzimas, em especial oxidorredutases.
Analisando os resultados do alinhamento múltiplo podemos ver, como de esperar pelos resultados do blast, que as sequências são bastante semelhantes entre si, exceto numa porção de aminoácidos existente na proteína em estudo e em duas outras proteínas. Nestas três, essa porção inicial de aminoácidos é igual, contudo, não existe nas restantes. A proteína em estudo não apresenta, no entanto, uma relação direta na árvore filogenética com nenhuma das sete proteínas que apresentam função conhecida. De facto, as proteínas encontram-se distantes da proteína em estudo e não é possível identificar um ancestral comum. Desta forma, não podemos inferir que a lpg0428 possui atividade de liase, apesar de muito provavelmente, devido ao domínio que possui, ser uma enzima.
 Figura 7 - Resultados do alinhamento múltiplo para a proteína lpg0428.
Figura 7 - Resultados do alinhamento múltiplo para a proteína lpg0428.
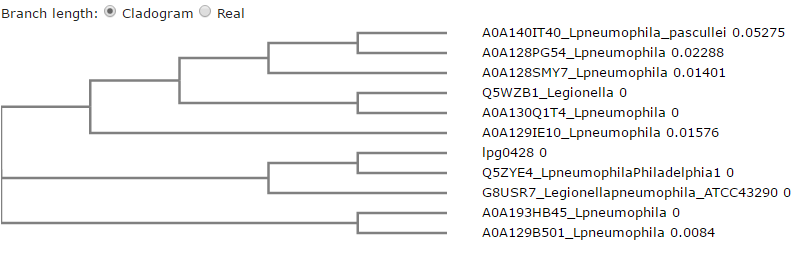 Figura 8 - Árvore filogenética para a proteína lpg0428.
Figura 8 - Árvore filogenética para a proteína lpg0428.