| lpg0233 |
[EC:4.1.1.7] |
- id: K01576 | descrição: benzoylformate decarboxylase
|
- id: lpn00627 | descrição: Aminobenzoate degradation
- id: lpn01120 | descrição: Microbial metabolism in diverse environments
|
- id: R01764 | descrição: phenylglyoxylate = benzaldehyde + CO2
|
| lpg0238 |
[EC:1.2.1.8] |
- id: K00130 | descrição: betaine-aldehyde dehydrogenase
|
- id: lpn00260 | descrição: Glycine, serine and threonine metabolism
- id: lpn01100 | descrição: Metabolic pathways
|
- id: R02565 | descrição: betaine aldehyde + NAD+ + H2O = betaine + NADH + 2 H+
|
| lpg0239 |
[EC:2.6.1.19|2.6.1.22] |
- id: K07250 | descrição: 4-aminobutyrate aminotransferase / (S)-3-amino-2-methylpropionate transaminase
|
- id: lpn00250 | descrição: Alanine, aspartate and glutamate metabolism
- id: lpn00280 | descrição: Valine, leucine and isoleucine degradation
- id: lpn00410 | descrição: beta-Alanine metabolism
- id: lpn00640 | descrição: Propanoate metabolism
- id: lpn00650 | descrição: Butanoate metabolism
- id: lpn01100 | descrição: Metabolic pathways
- id: lpn01120 | descrição: Microbial metabolism in diverse environments
|
- id: R01648 | descrição: 4-aminobutanoate + 2-oxoglutarate = succinate semialdehyde + L-glutamate
|
| lpg0241 |
[EC:3.5.1.2] |
- id: K01425 | descrição: glutaminase
|
- id: lpn00220 | descrição: Arginine biosynthesis
- id: lpn00250 | descrição: Alanine, aspartate and glutamate metabolism
- id: lpn00471 | descrição: D-Glutamine and D-glutamate metabolism
- id: lpn01100 | descrição: Metabolic pathways
|
- id: R00256 | descrição: L-glutamine + H2O = L-glutamate + NH3
|
| lpg0242 |
[EC:1.1.1.95] |
- id: K00058 | descrição: D-3-phosphoglycerate dehydrogenase
|
- id: lpn00260 | descrição: Glycine, serine and threonine metabolism
- id: lpn00680 | descrição: Methane metabolism
- id: lpn01100 | descrição: Metabolic pathways
- id: lpn01120 | descrição: Microbial metabolism in diverse environments
- id: lpn01130 | descrição: Biosynthesis of antibiotics
- id: lpn01200 | descrição: Carbon metabolism
- id: lpn01230 | descrição: Biosynthesis of amino acids
|
- id: R01513 | descrição: 3-phospho-D-glycerate + NAD+ = 3-phosphonooxypyruvate + NADH + H+
|
| lpg0244 |
[EC:1.16.1.1] |
- id: K00520 | descrição: mercuric reductase
|
|
- id: R02807 | descrição: Hg + NADP+ + H+ = Hg2+ + NADPH
|
| lpg0248 |
[EC:1.20.4.1] |
- id: K00537 | descrição: arsenate reductase
|
|
- id: R05747 | descrição: arsenate + glutaredoxin = arsenite + glutaredoxin disulfide + H2O
|
| lpg0249 |
[EC:1.14.19.1] |
- id: K00507 | descrição: stearoyl-CoA desaturase (Delta-9 desaturase)
|
- id: lpn01040 | descrição: Biosynthesis of unsaturated fatty acids
- id: lpn01212 | descrição: Fatty acid metabolism
|
- id: R02222 | descrição: stearoyl-CoA + 2 ferrocytochrome b5 + O2 + 2 H+ = oleoyl-CoA + 2 ferricytochrome b5 + 2 H2O
|
| lpg0270 |
[EC:6.3.4.21] |
- id: K00763 | descrição: nicotinate phosphoribosyltransferase
|
- id: lpn00760 | descrição: Nicotinate and nicotinamide metabolism
- id: lpn01100 | descrição: Metabolic pathways
|
- id: R01724 | descrição: nicotinate + 5-phospho-alpha-D-ribose 1-diphosphate + ATP + H2O = beta-nicotinate D-ribonucleotide + diphosphate + ADP + phosphate
|
| lpg0271 |
[EC:3.5.1.19| 3.5.1.-] |
- id: K08281 | descrição: nicotinamidase/pyrazinamidase
|
- id: lpn00760 | descrição: Nicotinate and nicotinamide metabolism
- id: lpn01100 | descrição: Metabolic pathways
|
- id: R01268 | descrição: nicotinamide + H2O = nicotinate + NH3
|
| lpg0283 |
[EC:1.2.1.2] |
- id: K00122 | descrição: formate dehydrogenase
|
- id: lpn00630 | descrição: Glyoxylate and dicarboxylate metabolism
- id: lpn00680 | descrição: Methane metabolism
- id: lpn01100 | descrição: Metabolic pathways
- id: lpn01120 | descrição: Microbial metabolism in diverse environments
- id: lpn01200 | descrição: Carbon metabolism
|
- id: R00519 | descrição: formate + NAD+ = CO2 + NADH
|
| lpg0288 |
[EC:5.4.3.-] |
- id: K19810 | descrição: L-lysine 2,3-aminomutase
|
|
|
| lpg0289 |
[EC:2.7.4.1] |
- id: K00937 | descrição: polyphosphate kinase
|
- id: lpn00190 | descrição: Oxidative phosphorylation
- id: lpn03018 | descrição: RNA degradation
|
- id: R02184 | descrição: ATP + (phosphate)n = ADP + (phosphate)n+1
|
| lpg0295 |
[EC:2.7.7.-] |
- id: K00992 | descrição: MurNAc alpha-1-phosphate uridylyltransferase
|
- id: lpn00520 | descrição: Amino sugar and nucleotide sugar metabolism
- id: lpn01100 | descrição: Metabolic pathways
|
|
| lpg0296 |
[EC:2.7.1.-] |
- id: K07102 | descrição: anomeric MurNAc/GlcNAc kinase
|
- id: lpn00520 | descrição: Amino sugar and nucleotide sugar metabolism
|
|
| lpg0299 |
[EC:1.1.1.262] |
- id: K00097 | descrição: 4-hydroxythreonine-4-phosphate dehydrogenase
|
- id: lpn00750 | descrição: Vitamin B6 metabolism
- id: lpn01100 | descrição: Metabolic pathways
|
- id: R05837 | descrição: 4-phosphonooxy-L-threonine + NAD+ = 3-amino-2-oxopropyl phosphate + CO2 + NADH + H+
|
| lpg0300 |
[EC:1.5.1.3] |
- id: K00287 | descrição: dihydrofolate reductase
|
- id: lpn00670 | descrição: One carbon pool by folate
- id: lpn00790 | descrição: Folate biosynthesis
- id: lpn01100 | descrição: Metabolic pathways
|
- id: R00939 | descrição: 5,6,7,8-tetrahydrofolate + NADP+ = 7,8-dihydrofolate + NADPH + H+
|
| lpg0361 |
[EC:2.3.1.41] |
- id: K00647 | descrição: 3-oxoacyl-[acyl-carrier-protein] synthase
|
- id: lpn00061 | descrição: Fatty acid biosynthesis
- id: lpn00780 | descrição: Biotin metabolism
- id: lpn01100 | descrição: Metabolic pathways
- id: lpn01212 | descrição: Fatty acid metabolism
|
- id: R02768 | descrição: an acyl-[acyl-carrier protein] + a malonyl-[acyl-carrier protein] = a 3-oxoacyl-[acyl-carrier protein] + CO2 + an [acyl-carrier protein]
|
| lpg0362 |
[EC:2.3.1.41] |
- id: K00647 | descrição: 3-oxoacyl-[acyl-carrier-protein] synthase
|
- id: lpn00061 | descrição: Fatty acid biosynthesis
- id: lpn00780 | descrição: Biotin metabolism
- id: lpn01100 | descrição: Metabolic pathways
- id: lpn01212 | descrição: Fatty acid metabolism
|
- id: R02768 | descrição: an acyl-[acyl-carrier protein] + a malonyl-[acyl-carrier protein] = a 3-oxoacyl-[acyl-carrier protein] + CO2 + an [acyl-carrier protein]
|
| lpg0363 |
[EC:2.3.1.241] |
- id: K02517 | descrição: Kdo2-lipid IVA lauroyltransferase
|
- id: lpn00540 | descrição: Lipopolysaccharide biosynthesis
- id: lpn01100 | descrição: Metabolic pathways
|
- id: R05146 | descrição: a dodecanoyl-[acyl-carrier protein] + Kdo2-lipid IVA = dodecanoyl-Kdo2-lipid IVA + an [acyl-carrier protein]
|
| lpg0366 |
[EC:5.1.1.7] |
- id: K01778 | descrição: diaminopimelate epimerase
|
- id: lpn00300 | descrição: Lysine biosynthesis
- id: lpn01100 | descrição: Metabolic pathways
- id: lpn01110 | descrição: Biosynthesis of secondary metabolites
- id: lpn01120 | descrição: Microbial metabolism in diverse environments
- id: lpn01130 | descrição: Biosynthesis of antibiotics
- id: lpn01230 | descrição: Biosynthesis of amino acids
|
- id: R02735 | descrição: LL-2,6-diaminoheptanedioate = meso-diaminoheptanedioate
|
| lpg0369 |
|
- id: K06999 | descrição: phospholipase/carboxylesterase
|
|
|
| lpg0409 |
|
- id: K14998 | descrição: surfeit locus 1 family protein
|
|
|
| lpg0411 |
|
- id: K02259 | descrição: cytochrome c oxidase assembly protein subunit 15
|
- id: lpn00190 | descrição: Oxidative phosphorylation
- id: lpn00860 | descrição: Porphyrin and chlorophyll metabolism
- id: lpn01100 | descrição: Metabolic pathways
- id: lpn01110 | descrição: Biosynthesis of secondary metabolites
- id: lpn02020 | descrição: Two-component system
|
|
| lpg0412 |
[EC:2.5.1.-] |
- id: K02301 | descrição: protoheme IX farnesyltransferase
|
- id: lpn00190 | descrição: Oxidative phosphorylation
- id: lpn00860 | descrição: Porphyrin and chlorophyll metabolism
- id: lpn01100 | descrição: Metabolic pathways
- id: lpn01110 | descrição: Biosynthesis of secondary metabolites
|
|
| lpg0416 |
[EC:1.1.1.49 | 1.1.1.363] |
- id: K00036 | descrição: glucose-6-phosphate 1-dehydrogenase
|
- id: lpn00030 | descrição: Pentose phosphate pathway
- id: lpn00480 | descrição: Glutathione metabolism
- id: lpn01100 | descrição: Metabolic pathways
- id: lpn01110 | descrição: Biosynthesis of secondary metabolites
- id: lpn01120 | descrição: Microbial metabolism in diverse environments
- id: lpn01130 | descrição: Biosynthesis of antibiotics
- id: lpn01200 | descrição: Carbon metabolism
|
- id: R00835 | descrição: D-glucose 6-phosphate + NADP+ = 6-phospho-D-glucono-1,5-lactone + NADPH + H+
|
| lpg0417 |
[EC:3.1.1.31] |
- id: K01057 | descrição: 6-phosphogluconolactonase
|
- id: lpn00030 | descrição: Pentose phosphate pathway
- id: lpn01100 | descrição: Metabolic pathways
- id: lpn01110 | descrição: Biosynthesis of secondary metabolites
- id: lpn01120 | descrição: Microbial metabolism in diverse environments
- id: lpn01130 | descrição: Biosynthesis of antibiotics
- id: lpn01200 | descrição: Carbon metabolism
|
- id: R02035 | descrição: 6-phospho-D-glucono-1,5-lactone + H2O = 6-phospho-D-gluconate
|
| lpg0418 |
[EC:4.2.1.12] |
- id: K01690 | descrição: phosphogluconate dehydratase
|
- id: lpn00030 | descrição: Pentose phosphate pathway
- id: lpn01100 | descrição: Metabolic pathways
- id: lpn01120 | descrição: Microbial metabolism in diverse environments
- id: lpn01200 | descrição: Carbon metabolism
|
- id: R02036 | descrição: 6-phospho-D-gluconate = 2-dehydro-3-deoxy-6-phospho-D-gluconate + H2O
|
| lpg0419 |
[EC:2.7.1.2] |
- id: K00845 | descrição: glucokinase
|
- id: lpn00010 | descrição: Glycolysis / Gluconeogenesis
- id: lpn00052 | descrição: Galactose metabolism
- id: lpn00500 | descrição: Starch and sucrose metabolism
- id: lpn00520 | descrição: Amino sugar and nucleotide sugar metabolism
- id: lpn00521 | descrição: Streptomycin biosynthesis
- id: lpn01100 | descrição: Metabolic pathways
- id: lpn01110 | descrição: Biosynthesis of secondary metabolites
- id: lpn01120 | descrição: Microbial metabolism in diverse environments
- id: lpn01130 | descrição: Biosynthesis of antibiotics
- id: lpn01200 | descrição: Carbon metabolism
|
- id: R00299 | descrição: ATP + D-glucose = ADP + D-glucose 6-phosphate
|
| lpg0420 |
[EC:4.1.2.14 | 4.1.3.42] |
- id: K01625 | descrição: 2-dehydro-3-deoxyphosphogluconate aldolase / (4S)-4-hydroxy-2-oxoglutarate aldolase
|
- id: lpn00030 | descrição: Pentose phosphate pathway
- id: lpn00630 | descrição: Glyoxylate and dicarboxylate metabolism
- id: lpn01100 | descrição: Metabolic pathways
- id: lpn01120 | descrição: Microbial metabolism in diverse environments
- id: lpn01200 | descrição: Carbon metabolism
|
- id: R05605 | descrição: 2-dehydro-3-deoxy-6-phospho-D-gluconate = pyruvate + D-glyceraldehyde 3-phosphate
|
| lpg0422 |
[EC:3.2.1.3] |
- id: K01178 | descrição: glucoamylase
|
- id: lpn00500 | descrição: Starch and sucrose metabolism
- id: lpn01100 | descrição: Metabolic pathways
|
- id: - | descrição: Hydrolysis of terminal (1->4)-linked alpha-D-glucose residues successively from non-reducing ends of the chains with release of beta-D-glucose
|
| lpg0425 |
[EC:4.99.1.1] |
- id: K01772 | descrição: ferrochelatase
|
- id: lpn00860 | descrição: Porphyrin and chlorophyll metabolism
- id: lpn01100 | descrição: Metabolic pathways
- id: lpn01110 | descrição: Biosynthesis of secondary metabolites
|
- id: R00310 | descrição: protoheme + 2 H+ = protoporphyrin + Fe2+
|
| lpg0460 |
[EC:2.1.2.3 | 3.5.4.10] |
- id: K00602 | descrição: phosphoribosylaminoimidazolecarboxamide formyltransferase / IMP cyclohydrolase
|
- id: lpn00230 | descrição: Purine metabolism
- id: lpn00670 | descrição: One carbon pool by folate
- id: lpn01100 | descrição: Metabolic pathways
- id: lpn01110 | descrição: Biosynthesis of secondary metabolites
- id: lpn01130 | descrição: Biosynthesis of antibiotics
|
- id: R04560 | descrição: 10-formyltetrahydrofolate + 5-amino-1-(5-phospho-D-ribosyl)imidazole-4-carboxamide = tetrahydrofolate + 5-formamido-1-(5-phospho-D-ribosyl)imidazole-4-carboxamide
|
| lpg0462 |
[EC:6.4.1.2 | 6.3.4.14] |
- id: K01961 | descrição: acetyl-CoA carboxylase, biotin carboxylase subunit
|
- id: lpn00061 | descrição: Fatty acid biosynthesis
- id: lpn01100 | descrição: Metabolic pathways
- id: lpn00620 | descrição: Pyruvate metabolism
- id: lpn00640 | descrição: Propanoate metabolism
- id: lpn01110 | descrição: Biosynthesis of secondary metabolites
- id: lpn01120 | descrição: Microbial metabolism in diverse environments
- id: lpn01130 | descrição: Biosynthesis of antibiotics
- id: lpn01200 | descrição: Carbon metabolism
- id: lpn01212 | descrição: Fatty acid metabolism
|
- id: R00742 | descrição: ATP + acetyl-CoA + HCO3- = ADP + phosphate + malonyl-CoA
|
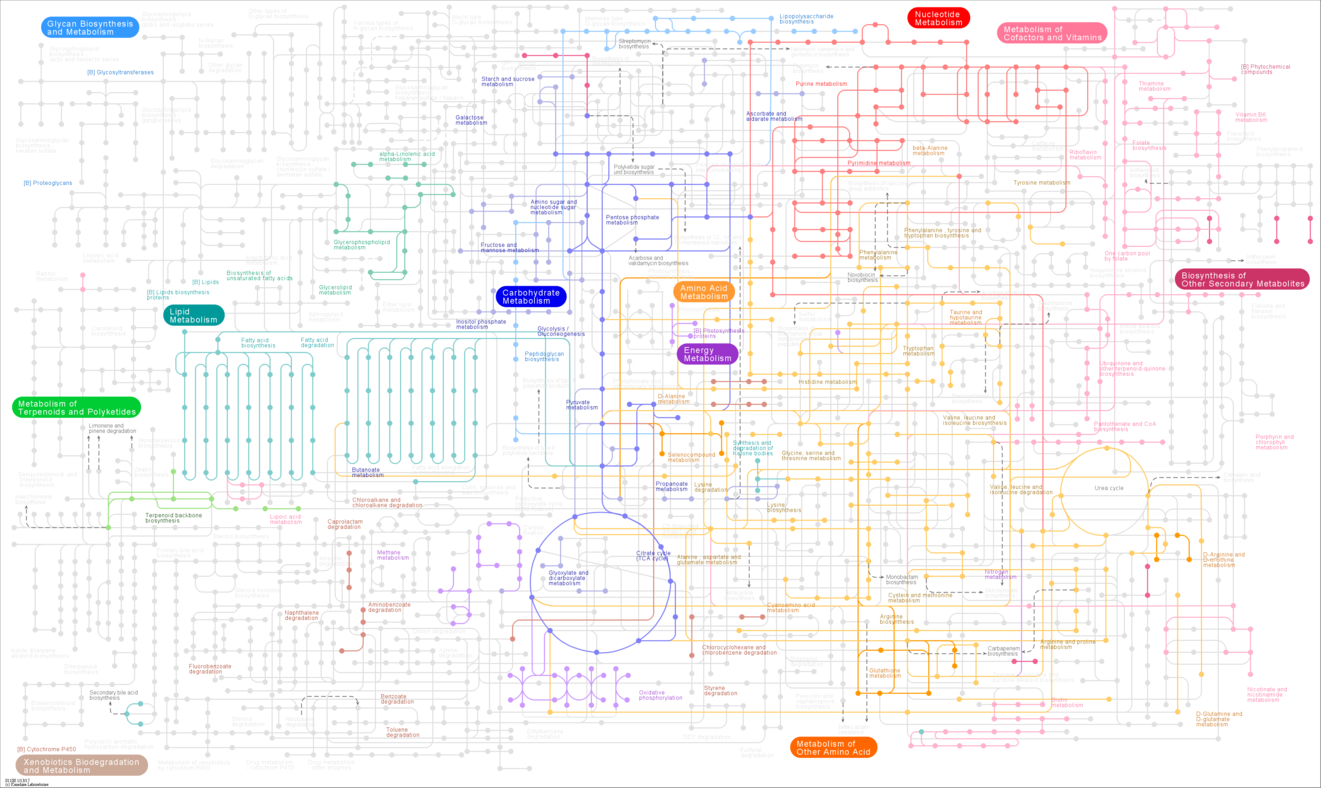 Figura1 - Rede Metabólica da Legionella Pneumophila susbp. pneumophila str. Philadelphia 1.
Figura1 - Rede Metabólica da Legionella Pneumophila susbp. pneumophila str. Philadelphia 1.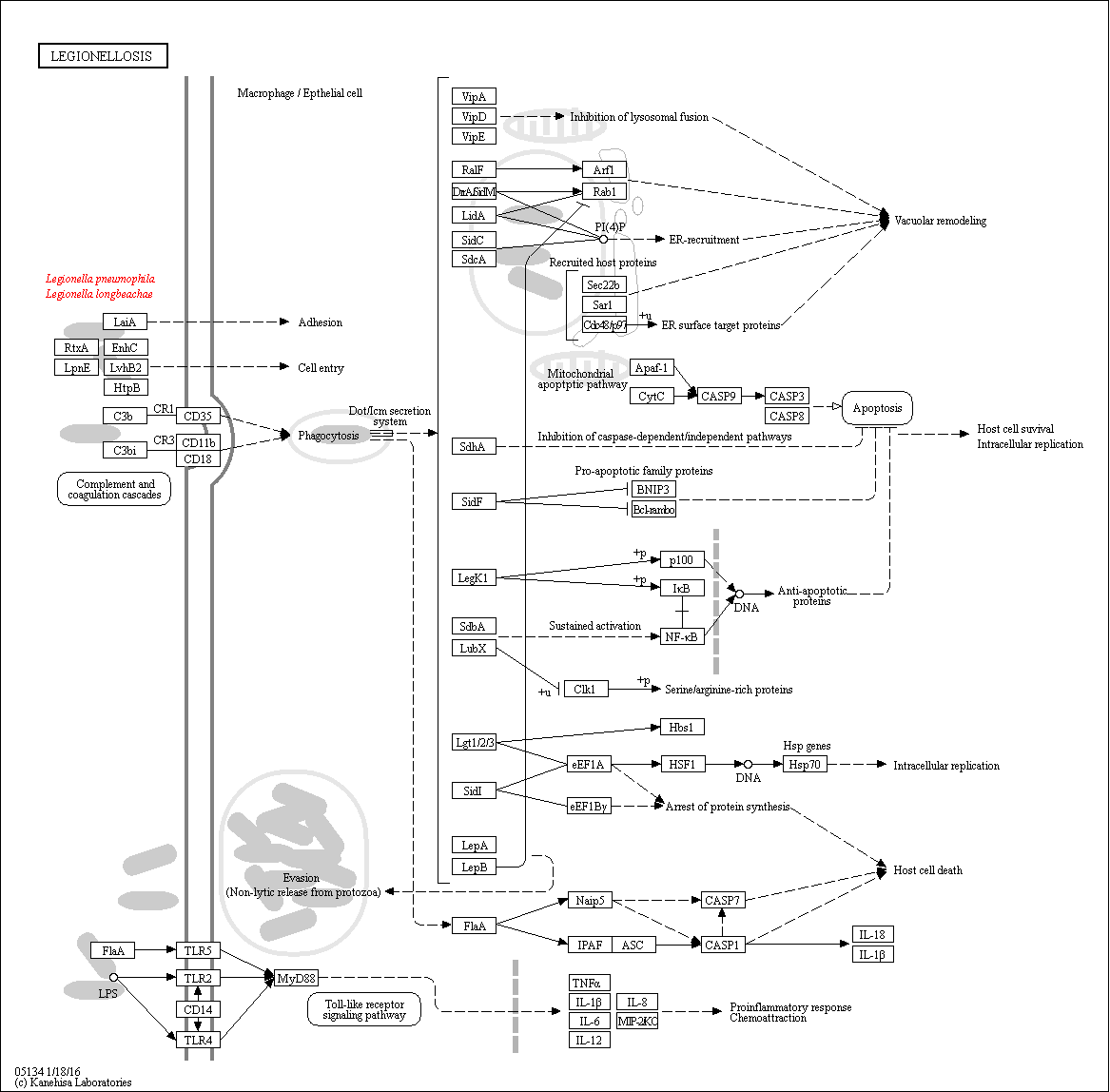 Figura 2 - Via responsável pela infeção provocada pela Legionella Pneumophila susbp. pneumophila str. Philadelphia 1.
Figura 2 - Via responsável pela infeção provocada pela Legionella Pneumophila susbp. pneumophila str. Philadelphia 1.