Legionella pneumophila subsp. pneumophila str. Philadelphia 1¶
A bactéria Legionella pneumophila é um bacilo facultativo pleomórfico gram-negativo aeróbico e mesófilo, com flagelo (indicador de mobilidade), não encapsulado, não formador de esporos e produtor de beta lactamases. Em termos de classificação filogenética pertence ao filo Proteobacteria e está enquadrada na classe Gammaproteobacteria, ordem Legionellales e família Legionellaceae. No meio ambiente é por norma encontrada em reservatórios aquáticos com temperaturas a rondar os 20ºC e 45ºC com depósitos de ferrugem, iodo e matéria orgânica, que favorecem o seu crescimento e multiplicação [1]. Existem cerca de 59 espécies/subespécies desta bactéria, e pelo menos 24 destas causam pneumonia humana [2].
Como é de esperar no que toca a patogéneos intracelulares, a análise do genoma da bactéria indica que a mesma sofreu transferência de genes horizontal, possuindo vários genes homólogos de eucariotas. Encontam-se ainda presentes sequências derivadas de fagos e sequências de inserção dispersas pelo genoma ou formando clusters. A estirpe Philadelphia 1 possui um elemento semelhante a um plasmídeo de 45kpb que existe como uma forma episomal circular ou no cromossoma [3]
Patogénese¶
A Legionella origina dois tipos de doenças a Febre de Pontiac e a Doença do legionário ou legionelose, sendo que a primeira se manifesta de forma menos severa enquanto a segunda pode chegar a causar pneumonias graves [4]. A patogénese da legionelose deve-se principalmente à capacidade da L. pneumophila de invadir e crescer dentro dos macrófagos alveolares. Depois de invadir o hospedeiro, a Legionella consegue evitar as atividades endossomais e lisossomais bactericidas e formar um fagossoma único e replicativo. É difícil fazer uma distinção entre os sintomas clínicos de ambas as patogenias e outras formas de pneumonia. Assim, a deteção da espécie de Legionella responsável por causar a doença é essencial para identificar infeções nosocomiais ou comunitárias, bem como para localizar a fonte da infeção. Os sistemas de secreção proteica tipo II e tipo IV são críticos para a patogénese da L. pneumophila. O sistema tipo II (Lps) segrega enzimas degradativas, enquanto o tipo IV (Dot/Icm) exporta proteínas efetoras que são essenciais para a formação e sobrevivência do fagossoma invadido pela legionella. A proteína prelina peptidase presente na membrana interna da bactéria, para além de facilitar a formação do pilus e a secreção tipo II, parece mediar uma terceira via operativa no hospedeiro humano [5,6]. Os genes responsáveis pela virulência (na zona do genoma 270001 a 505535) foram encontrados na base de dados Patrick: lpg0442; lpg0451; lpg0445; lpg0458; lpg0444; lpg0455; lpg0450; lpg0448; lpg0456; lpg0441; lpg0452; lpg0443; lpg0447 ;lpg0446; lpg0453; lpg0454; lpg0449; lpg0234; lpg0390; lpg0459; lpg0376; lpg0314; lpg0327.
Tratamento¶
De forma a combater a sua patogenia existem diversos fármacos a que esta bactéria é suscetível como a eritromicina, claritromicina, azitromicina, tetraciclina, moxifloxina e levofloxacina. Também os Aminoglicosídeos, como a gentamicina, kanamicina e estreptomicina, são ativos contra a Legionella pneumophila, contudo já existem estirpes mutantes que mostram grandes níveis de resistência à estreptomicina [5,6] e à rifampicina [7]. Através da base de dados Patrick identificamos os genes (na zona do genoma 270001 a 505535) responsáveis pela resistência a antibióticos, sendo que são os seguintes: lpg0314; lpg0322; lpg0327; lpg0429; lpg0430. Os genes que servem como alvos farmacológicos são os seguintes: lpg0328; lpg0314; lpg0327; lpg0324.
Os antibióticos de primeira linha para o tratamento de infeções por L. pneumophila incluem a azitromicina (da classe dos macrólidos) e o levoflaxin (da classe das fluroquinolonas). A azitromicina impede o crescimento da bactéria ao interferir com a sua síntese proteica. Este fármaco liga-se à subunidade 50S ribossomal, inibindo assim a tradução do mRNA.O levoflaxin inibe a síntese de DNA da bactéria, ao inibir a DNA girase e a topoisomerase IV, que desempenham funções na replicação e empacotamento do DNA [5,6].
Incidência¶
A doença do Legionário permanece incomum, maioritariamente esporádica e com baixos rácios de notificação nos países da União Europeia. 74% dos casos notificados distribuem-se por apenas 5 países, estando Portugal incluído. Em Vila France de Xira, Lisboa, ocorreu um outbreak de mais de 400 casos em Novembro de 2014. L. pneumophila serogrupo 1 foi o patogéneo mais comumente identificado, nomeadamente em 81% dos casos confirmados por cultura [8].
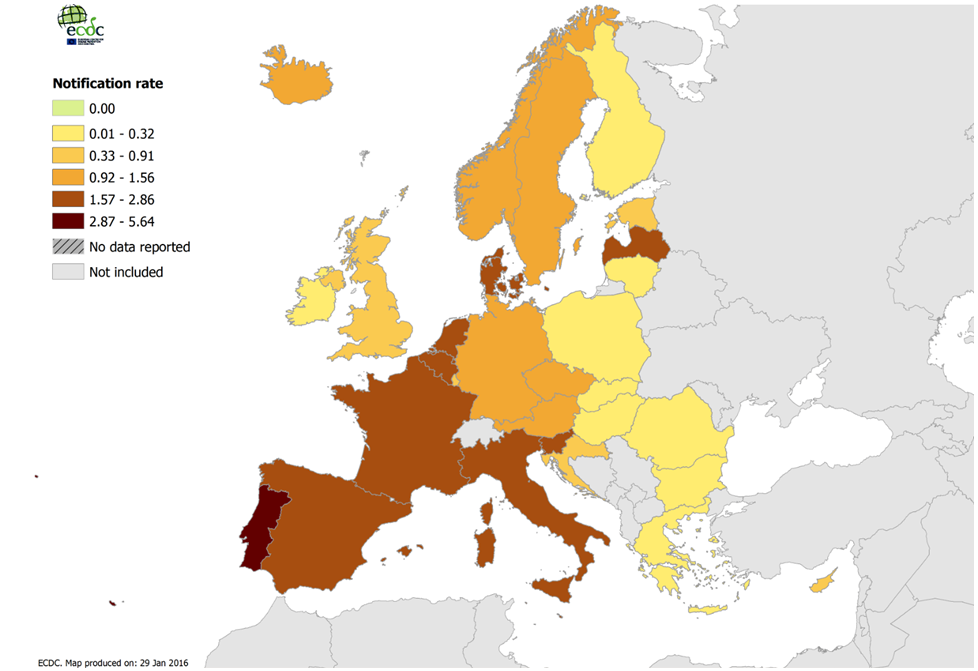 Figura 1 - Rácio dos casos da Doença do Legionario confirmados por cada 100 000 indivíduos por país, EU/EEA, 2014. Adaptado de (8).
Figura 1 - Rácio dos casos da Doença do Legionario confirmados por cada 100 000 indivíduos por país, EU/EEA, 2014. Adaptado de (8).
Iremos então proceder à analise da zona do genoma 270001 a 505535 da estirpe Legionella pneumophila subsp. pneumophila str. Philadelphia 1, recorrendo a bases de dados e ferramentas bioinformáticas. A estirpe em estudo apresenta um genoma circular, sendo que iremos abordar 200 genes dos aproximadamente 3000 existentes.
Referências Bibliográficas¶
1.NCBI [Internet]. Legionella pneumophila Causes Legionnaire’s disease. [cited 2017 Jan 15]. Available from: https://www.ncbi.nlm.nih.gov/genome/?term=Legionella+pneumophila+subsp.+pneumophila+str.+Philadelphia+1
2.Cho MS, Ahn TY, Joh K, Lee ES, Park DS. Improved PCR assay for the species-specific identification and quantitation of Legionella pneumophila in water. Appl Microbiol Biotechnol. 2015;99(21):9227–36.
3.Chien M. The Genomic Sequence of the Accidental Pathogen Legionella pneumophila. Science (80- ) [Internet]. 2004;305(5692):1966–8. Available from: http://www.sciencemag.org/cgi/doi/10.1126/science.1099776
4.Washington C. Winn J. Legionella. In: S B, editor. Medical Microbiology [Internet]. 4th ed. Galveston; 1996. Available from: https://www.ncbi.nlm.nih.gov/books/NBK7619/
5.Cianciotto NP. Pathogenicity of Legionella pneumophila. Int J Med Microbiol [Internet]. 2001;291(5):331–43.
6.Newton HJ, Ang DKY, Van Driel IR, Hartland EL. Molecular pathogenesis of infections caused by Legionella pneumophila. Clin Microbiol Rev. 2010;23(2):274–98
7.Moffie BG, Mouton RP. Sensitivity and resistance of Legionella pneumophila to some antibiotics and combinations of antibiotics. J Antimicrob Chemother. 1988;22(4):457–62.
8.European Centre for Disease Prevention and Control. Annual Epidemiological Report 2016 – Legionnaires' Disease. [Internet]. 2016 [cited 2017 01 15].